7月31日,吉林大学王国庆、周丰丰团队在国际顶级期刊《Briefings in Bioinformatics》(Q1, IF=9.10)上发表题为“Application of Bayesian phylogenetic inference modelling for evolutionary genetic analysis and dynamic changes in 2019-nCoV”的学术文章,应用计算生物学与分子生物学交叉学科技术对新型冠状病毒(2019-nCoV)的基因组修饰及分子进化特征进行分析。加拿大2.8预测在线预测100%正确博士生邵彤和硕士生王文芳是该论文的共同第一作者,计算机学院博士生段镁钰协助完成海量病毒数据的并行分析,药学院16级本科生刘宝玥通过大学生创新训练计划项目参与本项目的研究。加拿大2.8预测在线预测100%正确王国庆教授与计算机学院周丰丰教授为本文的共同通讯作者。加拿大2.8预测在线预测100%正确是该论文的第一完成单位。

2019-nCoV在全球范围内的感染病例已经超过1000万例,其传播速度之快给全球造成灾难性的损失。在该项研究中,研究人员利用贝叶斯方法构建了746株2019-nCoV的进化树,分析来自不同地区病毒株之间的进化关系。来自中国的毒株及美国毒株分属不同分支,且潜在修饰位点有所不同,提示不同地区的毒株会因为修饰的不同导致传染性和致病力上的差别。相比于SARS病毒,2019-nCoVORF6的相似度最低(69%),E蛋白具有最高的相似性(95%)。与来自蝙蝠的冠状病毒相比,则相似度最高的为E蛋白和ORF6(100%),相似度最低的是S蛋白(97%)。
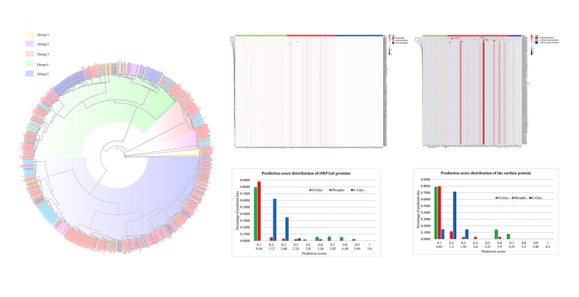
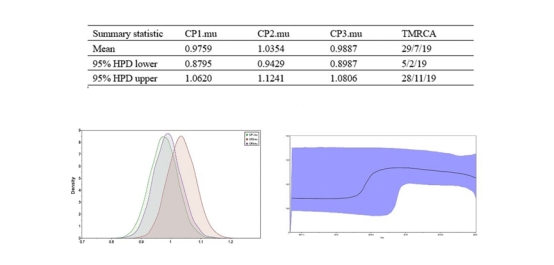
进化分子钟显示2019-nCoV共同祖先出现的时间早于报道的第一例病例。同时,利用贝叶斯天际线估测有效种群的活跃时间,其活跃时间也早于第一例发病病例,提示2019-nCoV或早就存在。该工作系统解析了2019-nCoV的传播方式及进化方向,对疾病的防治及预警具有重要的科学意义。
此研究得到了科技部“一带一路”传染病防治科技重大专项、国家自然科学基金和吉林省科技厅国际合作项目的支持。